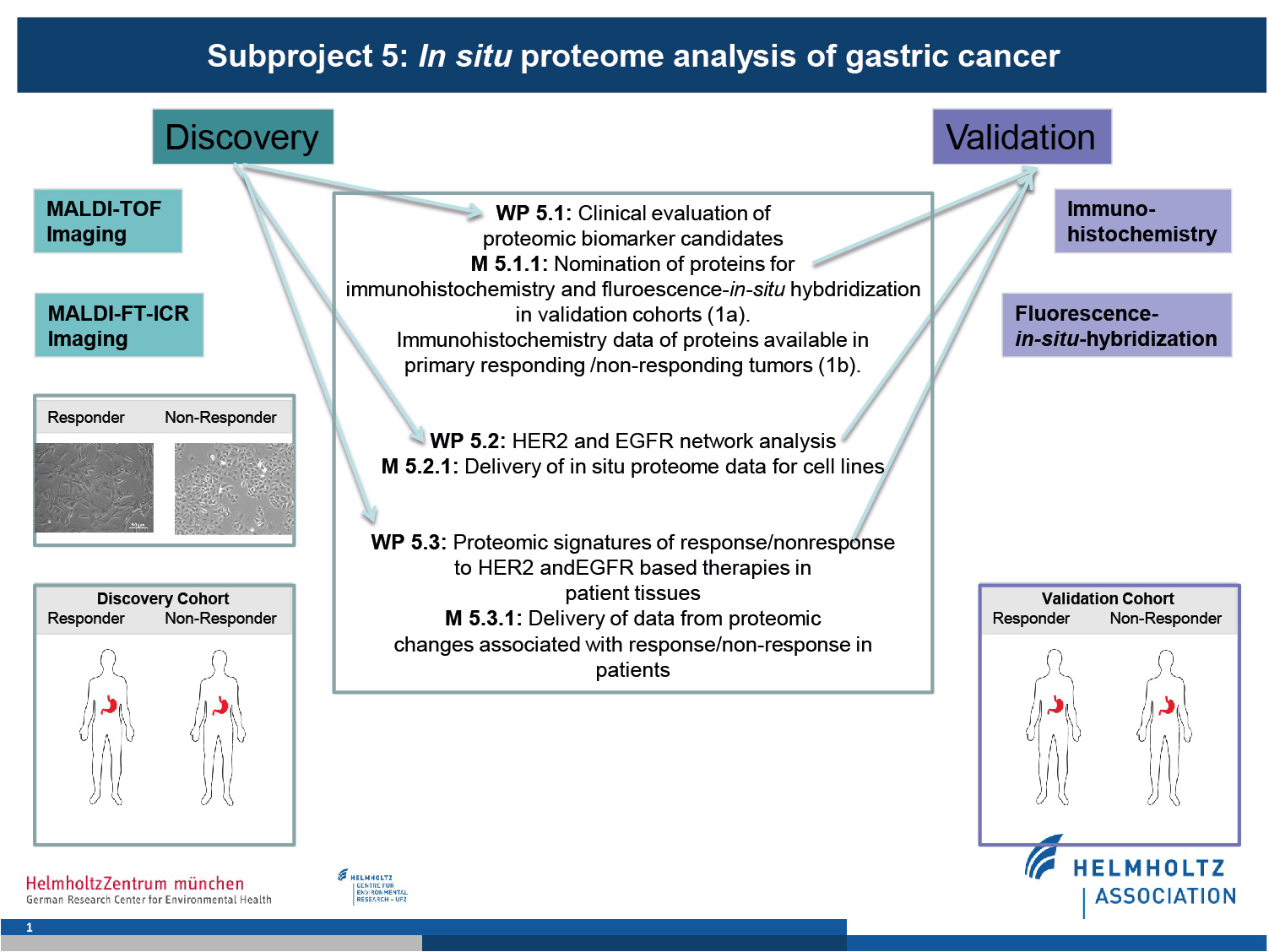
TP 5
In-situ Proteomanalyse des Magenkarzinoms
Veränderte Regulierung/Programmierung von Signalwegen und Netzwerken sind eine Ursache für die Tumorentwicklung und -resistenz. Human Epidermal Growth Factor 2 (HER2) und Epidermal Growth Factor Receptor (EGFR) sind Proteine, welche das Tumorwachstum und -überleben stimulieren. Klinische Studien mit neuen Medikamenten, die diese Rezeptoren und Signalwege zum Ziel haben, zeigten gemischte Reaktionen bei Patienten. TP 5 wird molekulare in situ-Analysen durchführen, um Mechanismen des Ansprechens/Resistenz gegen EGFR und HER2 gerichtete Therapien zu untersuchen.
Matrix-Assisted Laser Desorption/Ionisation Imaging (MALDI-MSI) ist eine massenspektrometrische Methode zur Untersuchung der Verteilung von Molekülen in situ. Im Gegensatz zu klassischen histologischen Methoden, ist MALDI-MSI in der Lage zahlreiche Moleküle in einer Probe gleichzeitig zu detektieren. MALDI-MSI zeigt die räumliche Verteilung von Molekülen auf – eine Information, die in der flüssigkeitsbasierten Massenspektrometrie verloren geht. Da es Hinweise auf molekulare Heterogenität von Magentumoren gibt, ist dies von Bedeutung. MALDI-MSI wird in diesem Projekt dazu verwendet Biomarkerkandidaten beim Magenkrebs zu identifizieren und zu evaluieren, und somit das Wissen über HER2 / EGFR-Signalwege zu erweitern.
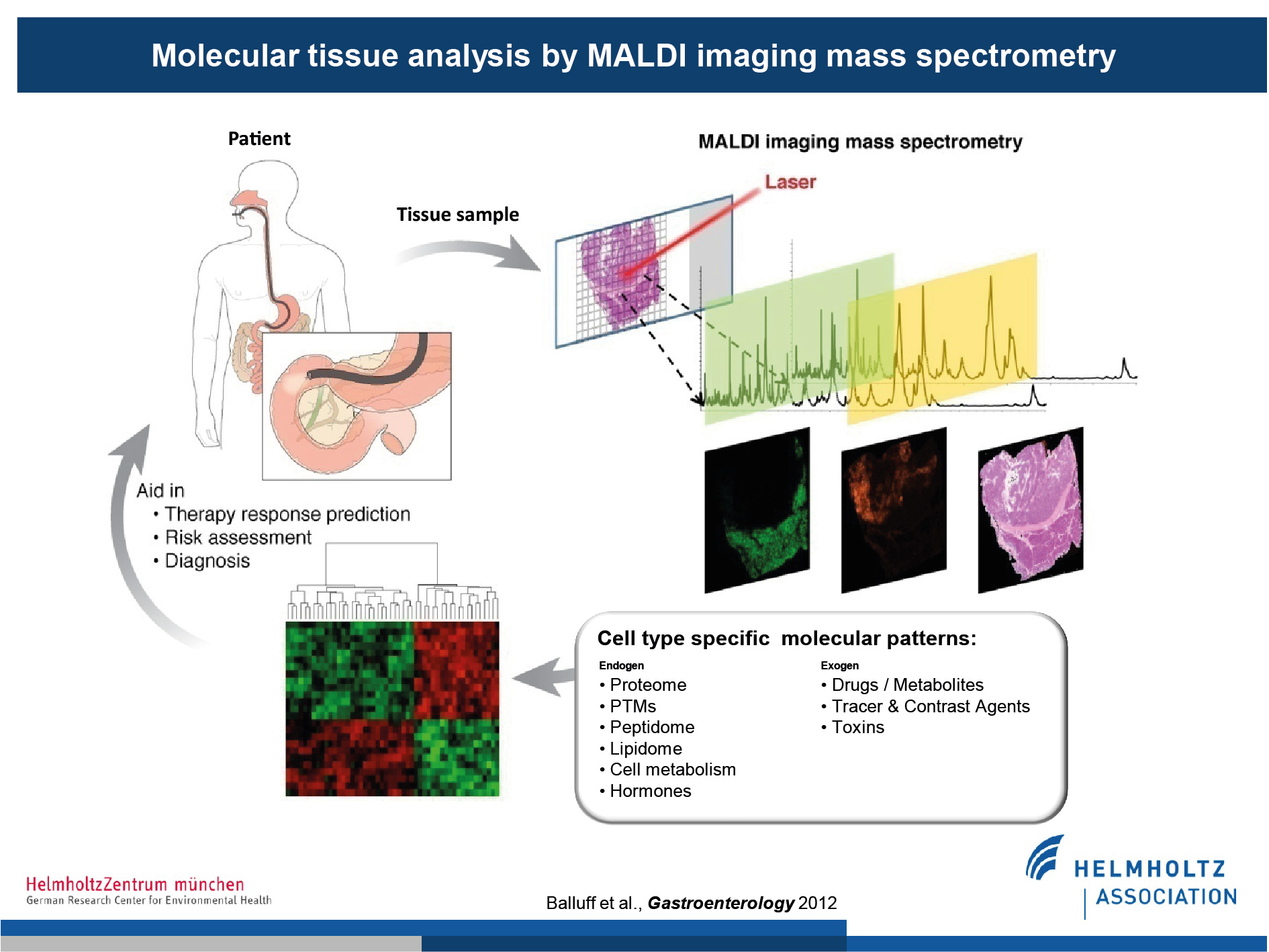
Patientenproben, wie Magenbiopsien, können mit MALDI-MSI gemessen werden. Die Messungen werden anschließend mit den korrespondierenden histologischen Schnitten koregistriert, was die Identifizierung von räumlich aufgelösten molekularen Signaturen erlaubt. Hierbei kann es sich um unterschiedlichste Molekülklassen handeln wie z.B. Proteine, Lipide, Wirkstoffe und deren Metaboliten welche wiederum Auskunft über klinisch relevante Fragestellungen wie die Vorhersage des Ansprechens oder der Resistenz auf eine Therapie geben können.
(Abbildung modifiziert von Balluff et al., Gastroenterology, 2012)
Das TP ist in drei Arbeitspakete aufgeteilt und stellt für das Konsortium wichtige Informationen.
1. Klinische Evaluation von proteomischen Biomarkerkandidaten
Mit MALDI-MSI wurden proteomische Profile von Magenkarzinomen erhoben, welche differenziert exprimierte Proteine zwischen individuellen Patienten aufgrund des HER2 und EGFR Status, der Tumorprogression und des Überlebens aufzeigten. Diese Daten werden SP2, SP3 und SP4 zur weiteren Identifizierung von Expressionsprofilen zur Verfügung gestellt, sowie Proteine herausgearbeitet welche ein Therapieansprechen oder –resistenz anzeigen.
2. HER2 und EGFR Netzwerkanalyse
Molekulare Profile von auf Therapie ansprechenden/resistenten Zellinien werden durch SP1 generiert. Diese Daten bilden die Grundlage für die Identifizierung von Proteinen für Netzwerkanalysen (SP3) und deren Verknüpfung in weitere krebsrelevante Signalwege.
3. Proteomische Signaturen des Ansprechens/Resistenz von Patienten auf HER2 und EGFR-basierte Therapien
Proben von Patienten aus klinischen Studien von SP6 werden mit MALDI-MSI vermessen und direkt verglichen und zu den Daten der Zelllinien von SP2, SP3 und SP4 integriert.
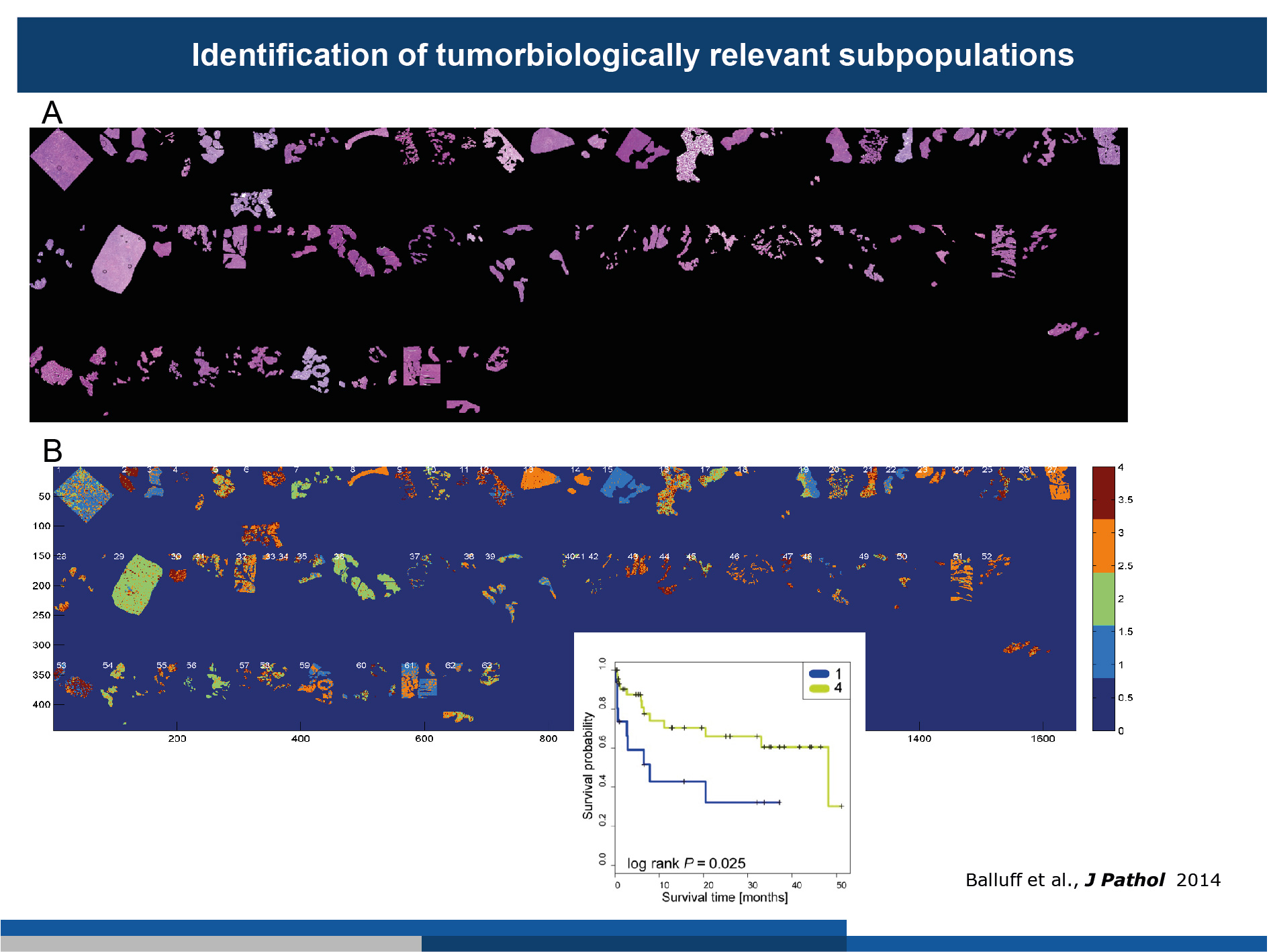
(A) 63 Magenkrebs-Patienten-Proben wurden mit MALDI-MSI vermessen. Die segmentierten Tumorzellen sind als Hämatoxylin und Eosin-Färbungen dargestellt.
(B) „Agreementanalysen“ ergaben eine substanzielle inter- und intratumorale Heterogenität. Durch die Korrelation von tumorsubpopulationen mit Überlebensdaten konnte spezifische Tumorsubpopulatoinen identifiziert werden welche mit dem Überleben von Patienten assoziiert sind.
(Abbildung modifiziert von Balluff et al., J Pathology, 2014)



